尹东、廖建友研究员团队通过单细胞扰动测序大规模分析基因功能和调控网络的工具平台-PerturbDB在《Nucleic Acids Research》杂志发表
论文题目:PerturbDB for unraveling gene functions and regulatory networks.
通讯作者:尹东;廖建友;林浩铭
第一作者:杨兵;张曼;时妍梅;郑冰琪
论文发表刊物:Nucleic Acids Research

【研究背景】
基因功能和调控网络的研究对于理解疾病的发生和发展机制至关重要,尤其是在癌症等复杂疾病中。尽管现有的基因敲除和敲降技术已经取得了一定的进展,但这些技术通常只能针对单个或少数基因进行研究,难以全面揭示基因网络的复杂性。Perturb-Seq技术结合了CRISPR基因编辑和单细胞RNA测序,能够在单个实验中同时分析数千个基因的转录组变化,为基因功能和调控网络的研究提供了新的机遇。本研究旨在开发一个用户友好的平台PerturbDB,帮助研究人员高效地利用Perturb-Seq数据集,揭示基因功能和调控网络。
【主要发现】
研究团队开发了PerturbDB平台,整合了66个Perturb-Seq数据集,涵盖了19种不同细胞系中10,194个基因的4,518,521个单细胞转录组。通过Mixscape算法统一处理所有数据集,研究者们鉴定出421个基因簇,其中157个在不同细胞环境中稳定。此外,通过整合化学扰动的转录组数据,研究者鉴定了552种潜在抑制剂,针对1,409个基因,包括实验验证的mTOR信号抑制剂视黄醇。
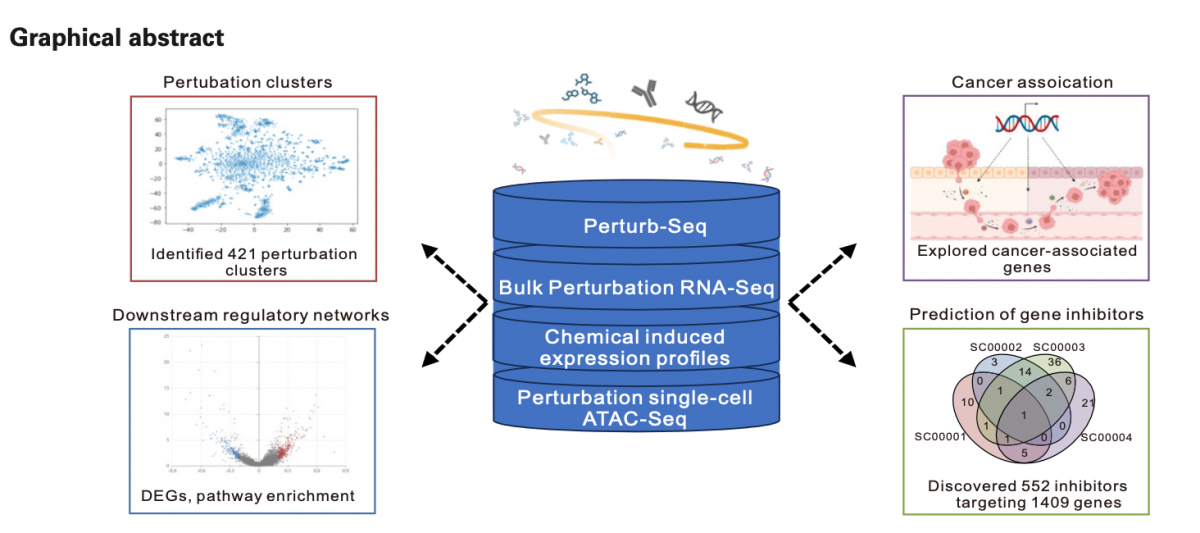
【研究意义】
PerturbDB平台为研究人员提供了一个宝贵的资源,用于探索基因功能和调控网络。该平台不仅有助于深入理解基因在癌症等疾病中的作用,还为开发新的治疗靶点和诊断标志物提供了理论基础。通过PerturbDB,研究人员可以轻松访问和分析大量的Perturb-Seq数据,推动对基因功能和疾病调控网络的进一步研究。
论文链接:https://academic.oup.com/nar/article/53/D1/D1120/7755478?login=false
阅读二维码:








 粤公网安备 44010402003003号
粤公网安备 44010402003003号 